| 标识符 | CSTR:16397.09.0B01001251 |
|---|---|
| 资源中文名称 | 调节肠道干细胞分化基因小鼠谱系示踪模型 |
| 资源英文名称 | Mouse lineage tracing model for testing the genes that regulate intestinal stem cell differentiation |
| 疾病概述 | 肠道干细胞位于肠粘膜隐窝基底部,即基底隐窝是肠道干细胞的细胞库。正常情况下,位于隐窝基底部的肠道干细胞不断向隐窝顶部(肠腔方向)迁移,整个迁移过程大约3-5d,在迁移过程中肠道干细胞分化形成不同的肠粘膜细胞。 |
| 实验动物背景信息 | 细胞因子激活酪氨酸蛋白激酶(JAK) /信号转导和转录活化因子(STAT) 5 是几乎所有肠道细胞因子,生长因子和生长激素下游转录活化因子 。去除或减少 STAT5 能够导致胚胎干细胞,造血干细胞,肝干细胞,髓样干细胞,以及乳腺干细胞分化异常和组织功能障碍 。 我们实验室报道了敲除肠上皮 STAT5 导致 Lgr5+ IESC 再生障碍,上皮屏障缺失,增加肠炎易感性 。相反, 短暂的 STAT5 酪氨酸磷酸化(pYSTAT5) 能够增加Lgr5+肠干细胞再生,保护肠道,减轻实验性肠炎。 但是, STAT5 是否能够调节 IESC 内源性的转录、激活因子,调节何种上游细胞因子以及诱导何种肠上皮分化,还未见报道。因此我们使用特定的 Stat5 复合基因工程小鼠与Lgr5CreER-GFP-RstdToamtoCreER 小鼠,进行肠上皮细胞分化的谱系追踪来确定持续性的 pYSTAT5 激活能够诱导肠道干细胞分化为何种细胞类型。 |
| 模型制作方法 | 实验动物:Lgr5-EGFP-CreERT2小鼠,ROSA26-tdTomato小鼠,Stat5 或 icS5 floxed小鼠 试剂:tamoxifen(100mg/kg),4%PFA 仪器:冰冻切片机,leica DMI8活细胞工作站 实验操作规程:将Lgr5-CreGFP;RstdToamto-CreER 小鼠系与Stat5 或 icS5 floxed小鼠杂交,分别为对照组(Lgr5-CreGFP;RstdToamto-CreER);实验1组(Lgr5-CreGFP;RstdToamto-CreER;Stat5);实验2组(Lgr5-CreGFP;RstdToamto-CreER;icS5)。在小鼠4周龄时,提取杂交小鼠鼠尾DNA,通过凝胶电泳检测小鼠基因型。Lgr5基因PCR引物序列如下(扩增目的片段长174bp)。Common 8060:5'-CTGCTCTCTGCTCCCA GTCT-3';Wild Type 8061:5'-ATACCCCATCCCTT TTGAGC-3';Mutant 9402:5'-GAACTTCAGGGTC AGCTTGC-3'。PCR条件:预变性94℃3min;94℃变性30s,66℃复性1min,72℃延伸30s,35个循环;72℃延伸5min。选取双阳性小鼠,6~8周龄,体重20~22g。实验组小鼠按100mg/kg体重腹腔注射他莫昔芬,对照组小鼠注射等量的玉米油。通过 Tam 诱导, 然后分别在 12 小时和 1, 3, 5, 7, 30, 122和280天麻醉处死各组小鼠。分别取结肠、空肠、回肠组织,用冷PBS冲洗肠道组织,纵向剖开,放入多聚甲醛中固定过夜,制作冰冻切片在子代肠上皮细胞中追踪检测是否去除 Stat5 或活化 STAT5 能够改变肠上皮中的tdToamto 信号强度和位置. |
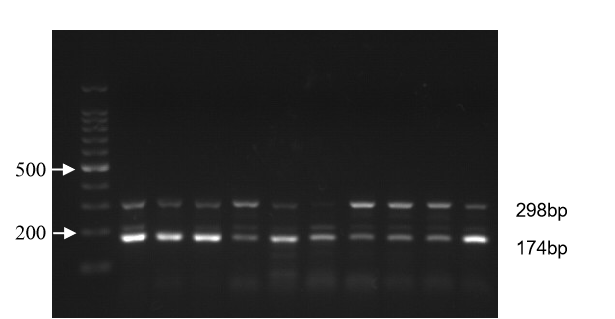
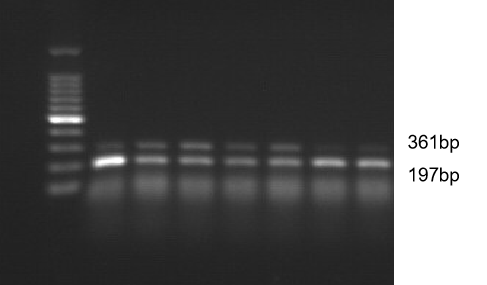
| 模型表型数据 | 1、 基因鉴定信息
图1 Lgr5, Rosa26tdTomato基因凝胶电泳鉴定结果 2.组织化学染色结果
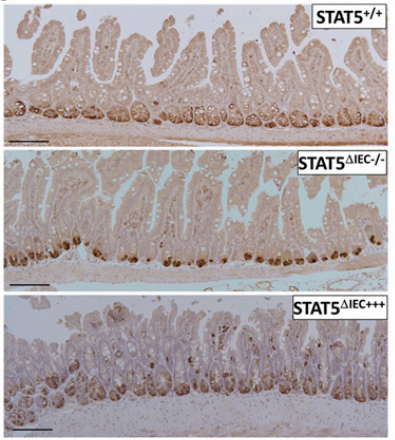
图2 潘氏细胞免疫组织化学染色结果
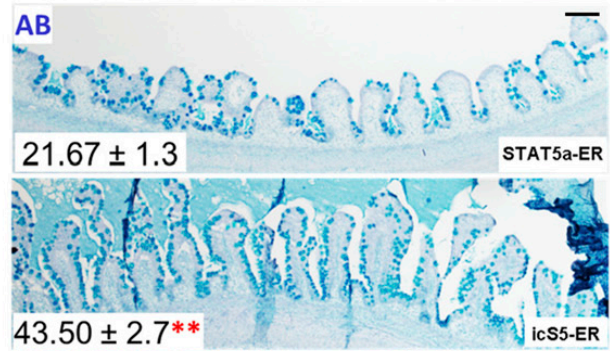
图3 杯状细胞染色结果 3.谱系追踪系统的建立
|
| 动物模型的评价与验证 | 在显微镜下观察诱导后不同时间点小肠干细胞的变化,发现诱导后3d隐窝底部已经开始出现荧光,并有向上移动的趋势; 诱导后5d荧光细胞已经移动到隐窝的快速增殖细胞(TA)部; 诱导后7d可看到有少量从隐窝分裂分化的细胞已经移动到绒毛顶端;诱导后14d有更多的荧光细胞到达小肠绒毛顶端,少量绒毛从隐窝基底到绒毛顶尖则全部带有荧光;诱导后45d可见整个小肠绒毛中荧光细胞更多、 更密实。 提示分化的细胞系来源于Lgr5+干细胞, 小鼠小肠干细胞谱系追踪模型成功建立。 |
| 保存方式 | 冷冻 |
| 合作方式 | 仅限合作研究 |
| 相关文章 | |
| 备注 |



 京公网安备 11010502043083号
京公网安备 11010502043083号